 Una parte importante del genoma de un organismo es no codificante; es decir, no lleva información para hacer proteínas. Parte del ADN no codificante se transcribe a moléculas funcionales de ARN no codificante (por ejemplo, ARN de transferencia, ARN ribosómico y ARN reguladores). Otras funciones del ADN no codificante son la regulación transcripcional y traslacional de las secuencias codificantes de proteínas, las regiones de unión al andamiaje nuclear, los orígenes de replicación del ADN, los centrómeros y los telómeros.
Una parte importante del genoma de un organismo es no codificante; es decir, no lleva información para hacer proteínas. Parte del ADN no codificante se transcribe a moléculas funcionales de ARN no codificante (por ejemplo, ARN de transferencia, ARN ribosómico y ARN reguladores). Otras funciones del ADN no codificante son la regulación transcripcional y traslacional de las secuencias codificantes de proteínas, las regiones de unión al andamiaje nuclear, los orígenes de replicación del ADN, los centrómeros y los telómeros.
El ADN codificante es aproximadamente el 2% en la especie humana, mientras que el genoma no codificante es el 98% restante.  Es un error denominar al ADN no codificante como ADN basura porque está implicado en la actividad epigenética y en complejas redes de interacciones genéticas, en palabras sencillas, en cuándo, dónde y cuánto se expresan los genes.
Es un error denominar al ADN no codificante como ADN basura porque está implicado en la actividad epigenética y en complejas redes de interacciones genéticas, en palabras sencillas, en cuándo, dónde y cuánto se expresan los genes.
A diferencia de los segmentos codificantes (exones), los límites de las regiones no codificantes no están bien definidos y, por lo tanto, diferentes tipos de marcas como la conservación evolutiva, la restricción de la secuencia y las marcas epigenéticas son guías útiles para su identificación y análisis.
 Un estudio reciente cuyo primer autor es Evin Padhi ha demostrado que hay mutaciones espontáneas en las regiones no codificantes del genoma que están relacionadas con el autismo. Por tanto, la genética del autismo no se limita a los genes codificantes sino que el ADN no codificante también influye y puede explicar el autismo de algunas personas afectadas.
Un estudio reciente cuyo primer autor es Evin Padhi ha demostrado que hay mutaciones espontáneas en las regiones no codificantes del genoma que están relacionadas con el autismo. Por tanto, la genética del autismo no se limita a los genes codificantes sino que el ADN no codificante también influye y puede explicar el autismo de algunas personas afectadas.
Los estudios estadísticos habían sugerido que los segmentos no codificantes presentaban variantes que contribuían al autismo y este estudio es el primero que ha identificado tres personas con mutaciones en el genoma no codificante. Parece claro que en el futuro cercano habrá una lista bastante amplia de variantes de novo en las secuencias no codificantes implicadas en el autismo, al igual que lo tenemos ya en el ADN codificante.
Los investigadores realizaron la secuenciación del genoma completo en 2671 familias con autismo (cohorte de descubrimiento de 516 familias, cohorte de replicación de 2155 familias). Centraron el estudio en 544 segmentos no codificantes del genoma que se sabe están activos en el cerebro de embriones de ratón. 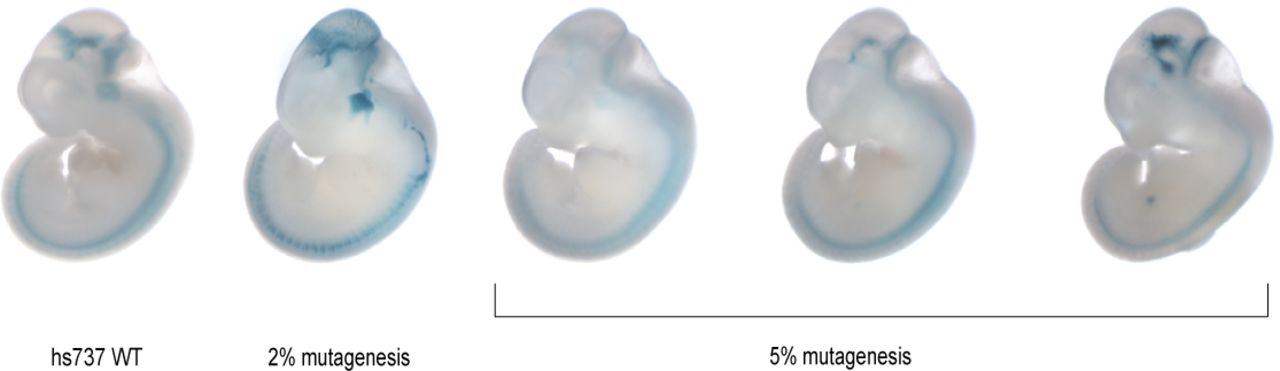 Estos segmentos, denominados potenciadores, aumentan la transcripción de los genes codificantes.
Estos segmentos, denominados potenciadores, aumentan la transcripción de los genes codificantes.
A continuación, buscaron en las secuencias del genoma completo de las 2.671 familias con un hijo autista, presentes en la base de datos de la Colección Simons Simplex, para ver cuáles de estos potenciadores contenían variantes de novo. Es decir, primero identificaron potenciadores que actúan en el cerebro y luego buscaron en el ADN no codificante de proteínas pero que codifica los potenciadores, si había diferencias entre las familias con autismo y las familias sin autismo.
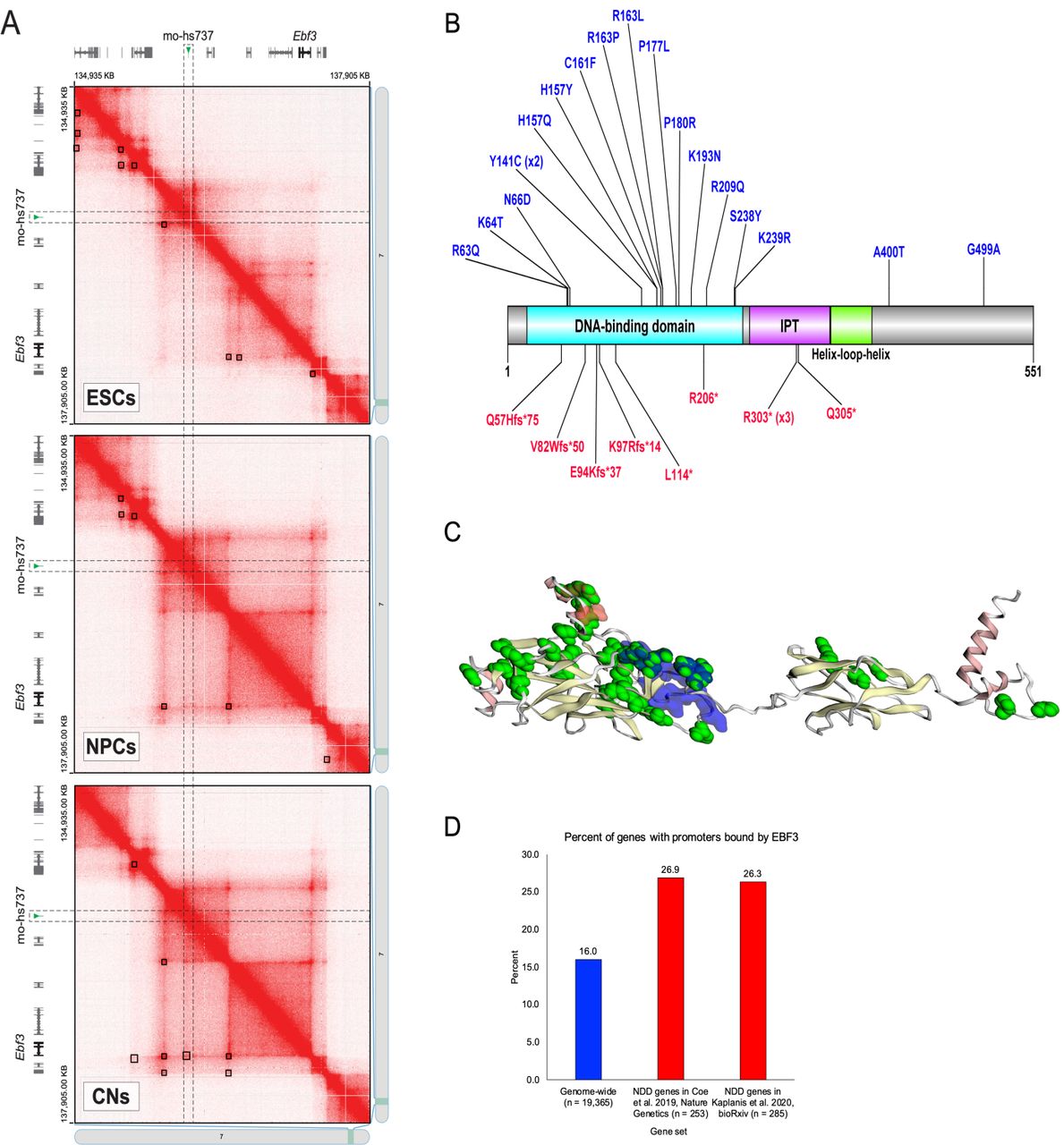 Un potenciador en particular, hs737, mostró un número significativamente mayor de variaciones de novo tanto en la cohorte de descubrimiento de 516 familias como en la cohorte de replicación de 2.155 familias. Cabe destacar que estas mutaciones de novo en hs737 aparecieron en tres personas con autismo sin otras variantes conocidas relacionadas con el autismo; es decir, personas con autismo que no presentaban genes codificantes relacionados con el autismo sí pueden presentar ADN no codificante relacionado con el autismo.
Un potenciador en particular, hs737, mostró un número significativamente mayor de variaciones de novo tanto en la cohorte de descubrimiento de 516 familias como en la cohorte de replicación de 2.155 familias. Cabe destacar que estas mutaciones de novo en hs737 aparecieron en tres personas con autismo sin otras variantes conocidas relacionadas con el autismo; es decir, personas con autismo que no presentaban genes codificantes relacionados con el autismo sí pueden presentar ADN no codificante relacionado con el autismo.
Los individuos con variaciones de novo en hs737 tenían fenotipos compartidos, coincidían en ser varón, función cognitiva intacta, e hipotonía o retraso motor. Esto contrasta con las variaciones de novo en los genes codificantes, que son más abundantes en mujeres con discapacidad intelectual. Usando una línea celular neuronal, los investigadores vieron que en los tres casos, las mutaciones de novo hicieron que el potenciador redujera, y no aumentara, la expresión génica. Mediante análisis epigenómicos, los investigadores pudieron ver que el hs737 es específico del cerebro y actúa sobre el gen del factor de transcripción EBF3 en el cerebro fetal humano.
Para sorpresa de los investigadores, el EBF3 es un gen bien estudiado. Las mutaciones en el EBF3 causan el síndrome de hipotonía, ataxia y retraso en el desarrollo (HADDS), caracterizado por un bajo tono muscular y problemas motores.  EBF3 presenta variaciones de novo en trastornos del neurodesarrollo y se expresa ampliamente en el cuerpo. El resultado no es similar en los casos en que la variación de novo (VDN) relacionada con EBF3 está en zonas codificantes o en no codificantes. Los individuos con VDNs codificantes tienen una mayor gravedad fenotípica (hipotonía, ataxia y síndrome de desarrollo retardado) en comparación con los individuos con variaciones de novo no codificantes que tienen autismo e hipotonía.
EBF3 presenta variaciones de novo en trastornos del neurodesarrollo y se expresa ampliamente en el cuerpo. El resultado no es similar en los casos en que la variación de novo (VDN) relacionada con EBF3 está en zonas codificantes o en no codificantes. Los individuos con VDNs codificantes tienen una mayor gravedad fenotípica (hipotonía, ataxia y síndrome de desarrollo retardado) en comparación con los individuos con variaciones de novo no codificantes que tienen autismo e hipotonía.
Los investigadores analizaron la unión de EBF3 a promotores e identificaron 3100 genes (16% de todos los genes del genoma) a los que se une EBF3, una cantidad asombrosa. A continuación, se centraron en los genes que codifican VDNs en trastornos del neurodesarrollo y a los que se unía EBF3. De los 253 genes implicados en trastornos de neurodesarrollo descritos por Coe et al. (2015), el 26,9% de ellos presentaban unión de EBF3 a su promotor. Estos VDNs se encuentran en nucleótidos altamente conservados y se cree que afectan a la unión de los factores de transcripción con el potenciador.
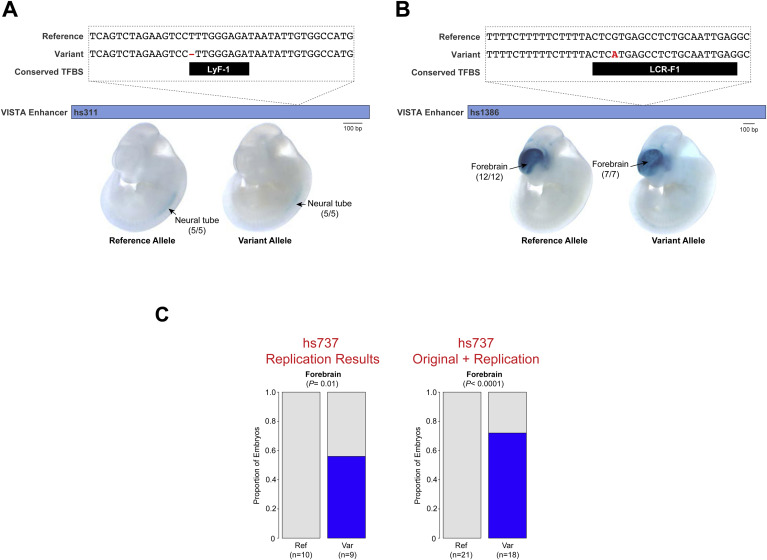 El hs737 sólo está activo en el cerebro mientras el embrión se está desarrollando, así que las mutaciones en el potenciador podrían dar lugar a rasgos similares pero menos graves que las mutaciones en el propio gen. En otras palabras, si afectas a un gen codificador el efecto puede ser total, si afectas al potenciador de ese gen el efecto podría ser parcial y no tan grave. Eso explicaría también la presencia del espectro: las mutaciones de un gen codificante pueden hacer que sea funcional o no, las mutaciones de un ADN no codificante (regulador) pueden hacer teóricamente que un gen se exprese al 30, 70 o 150% de lo normal.
El hs737 sólo está activo en el cerebro mientras el embrión se está desarrollando, así que las mutaciones en el potenciador podrían dar lugar a rasgos similares pero menos graves que las mutaciones en el propio gen. En otras palabras, si afectas a un gen codificador el efecto puede ser total, si afectas al potenciador de ese gen el efecto podría ser parcial y no tan grave. Eso explicaría también la presencia del espectro: las mutaciones de un gen codificante pueden hacer que sea funcional o no, las mutaciones de un ADN no codificante (regulador) pueden hacer teóricamente que un gen se exprese al 30, 70 o 150% de lo normal.
Un problema clave en el estudio de los potenciadores es determinar qué gen regulan. Es especialmente complicado cuando los potenciadores están muy alejados del promotor al que se dirigen, pero hay trucos para conseguirlo. El EBF3 es un factor de transcripción que se expresa de forma ubicua en los seres humanos, afecta a muchos genes, en particular en las células neuronales.  Los genes reguladores forman una amplia red en el que unos regulan a otros, en mecanismos en cascada. Cuanto más alta es la ubicación de la variante en la red, más grave es la alteración fenotípica, hay muchos genes alterados, y cuanto más abajo en la cascada, menos grave es la consecuencia fenotípica (por ejemplo, la mutación en el potenciador hs737). EBF3 regula otros genes implicados en la regulación posterior en trastornos del neurodesarrollo.
Los genes reguladores forman una amplia red en el que unos regulan a otros, en mecanismos en cascada. Cuanto más alta es la ubicación de la variante en la red, más grave es la alteración fenotípica, hay muchos genes alterados, y cuanto más abajo en la cascada, menos grave es la consecuencia fenotípica (por ejemplo, la mutación en el potenciador hs737). EBF3 regula otros genes implicados en la regulación posterior en trastornos del neurodesarrollo.
Los investigadores probaron muchas secuencias, pero solo un potenciador alcanzó una diferencia que entra dentro del nivel de significación establecido en este estudio. Sin embargo, parece evidente que se irán encontrando otros reguladores en los que también se verá una relación con el autismo. Finalmente, el grupo de Padhi está trabajando en el desarrollo de una cepa de ratones que carece del potenciador hs737, con el fin de comprender mejor cómo las mutaciones en esa secuencia de ADN no codificante afectan al comportamiento.
Para leer más:
- Coe BP, Stessman HAF, Sulovari A, Geisheker MR, Bakken TE, Lake AM, Dougherty JD, Lein ES, Hormozdiari F, Bernier RA, Eichler EE (2019) Neurodevelopmental disease genes implicated by de novo mutation and copy number variation morbidity. Nat Genet51(1): 106–116. doi: 10.1038/s41588-018-0288-4.
- Padhi EM, Hayeck TJ, Cheng Z, Chatterjee S, Mannion BJ, Byrska-Bishop M, Willems M, Pinson L, Redon S, Benech C, Uguen K, Audebert-Bellanger S, Le Marechal C, Férec C, Efthymiou S, Rahman F, Maqbool S, Maroofian R, Houlden H, Musunuri R, Narzisi G, Abhyankar A, Hunter RD, Akiyama J, Fries LE, Ng JK, Mehinovic E, Stong N, Allen AS, Dickel DE, Bernier RA, Gorkin DU, Pennacchio LA, Zody MC, Turner TN (2021) Coding and noncoding variants in EBF3 are involved in HADDS and simplex autism. Hum Genomics 15(1): 44. doi: 10.1186/s40246-021-00342-3.
- Schenkman L (2021) Mutations in the noncoding genome contribute to autism. Spectrum 2 de Agosto. https://www.spectrumnews.org/news/mutations-in-the-noncoding-genome-contribute-to-autism/ https://doi.org/10.53053/VCFT1127


Responder a RAFAEL GARCIA MONTEROCancelar respuesta